2025年9月,国际顶级期刊《Nature Methods》发表了来自北京昌平实验室、北京大学前沿交叉学科研究院等团队的重磅研究成果——蛋白质复合物结构预测模型GRASP。这项研究由高毅勤教授团队领衔,联合北京大学生命科学学院、化学与分子工程学院及华为技术有限公司昇思MindSpore AI4S团队共同完成,论文的发表标志着中国科学家在AI+生物计算领域取得突破性新进展。
论文地址:
nature.com/articles/s41592-025-02820-1
该论文提出的GRASP模型通过直接整合多种实验约束信息,显著提高了蛋白质复合物结构预测的准确性和可靠性。GRASP在处理模拟和真实世界的实验约束方面表现出色,特别是在抗体-抗原复合物预测中超越了现有的AlphaFold3。
GRASP还可以作为综合建模工具,结合多种实验信息进行复杂的约束建模。未来的工作将进一步优化GRASP的集成策略,以解决更具挑战性的应用场景。
# 01
核心创新:实验数据与AI的深度耦合
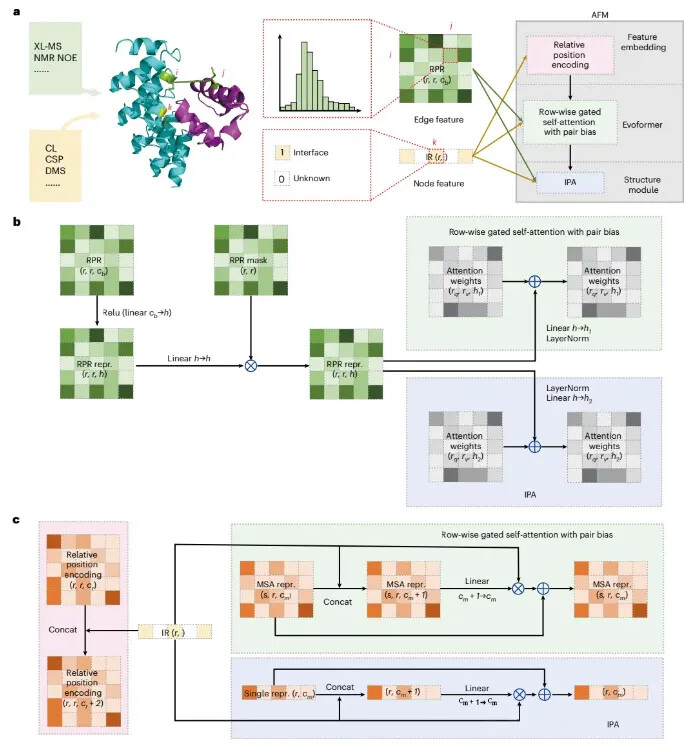
GRASP的核心突破在于首创"实验约束引导AI预测"的范式:
- 数据转化技术:将交联质谱(XL-MS)、深度突变扫描(DMS)等稀疏实验数据转化为残基对限制(RPR)和界面限制(IR),通过修改AlphaFold-Multimer的注意力机制实现精准嵌入 。
- 抗噪系统:开发迭代噪声过滤算法,可自动识别并剔除错误实验数据(如误差超过30%的XL-MS数据) 。
- 多模态融合架构:支持冷冻电镜密度图、化学位移扰动等6类实验数据混合输入,首次实现"低分辨率实验+高精度AI"的协同建模 。
# 02
部分创新场景性能倍级领先AlphaFold3
GRASP 在部分公开测试中表现优异:
- 在界面基准测试中,GRASP方法表现全面最佳。其效率极高,仅需引入两个跨链接触约束,就能将DockQ评分提升至0.35,并使过半样本达到可接受水平。
- GRASP在XL、CL、CSP约束数据实验中,对低覆盖率或高噪声的各类实验数据中均表现稳健,性能显著优于其他方法。
- 在抗原-抗体预测上,GRASP在深度突变扫描数据中展现出卓越性能与强鲁棒性,预测成功率与精度均大幅领先AlphaFold3结果23%-38%。
这项研究不仅突破了AlphaFold系列的部分技术瓶颈,也丰富了实验科学与AI协同的新范式,未来将有望在生物医药领域取得重要应用。